干货丨教你如何分析CRISPR文库数据找到靶点!

CRISPR文库筛选是一种基于CRISPR/Cas9系统发展而来的高通量基因筛选方法:在构建完成涵盖有多条不同sgRNA的文库细胞cell pool后,使用特定的筛选条件富集靶细胞,并结合NGS测序以及生信分析的方法,找出表型相关靶点基因。许多小伙伴可能已经掌握了CRISPR文库筛选的原理以及流程/方法,然而对筛选后的结果分析、靶点寻找仍然存有大量疑问。为了解答大家的疑问,今天,小源就带大家系统性地了解一下CRISPR文库分析的流程并解答一下大家的问题吧。
CRISPR文库分析流程
1.测序数据质控
NGS测序所得到的原始测序文件(raw reads)中,含有部分带接头、低质量的reads,为保证分析质量,需要先对raw reads进行过滤,得到clean reads。随后还需要根据Q20、Q30评估测序数据质量,通常,Q20 > 90% 或Q30 > 85% (图1)认为测序数据质量合格,若低于该数值说明测序质量低,数据误差大,需重新进行测序。
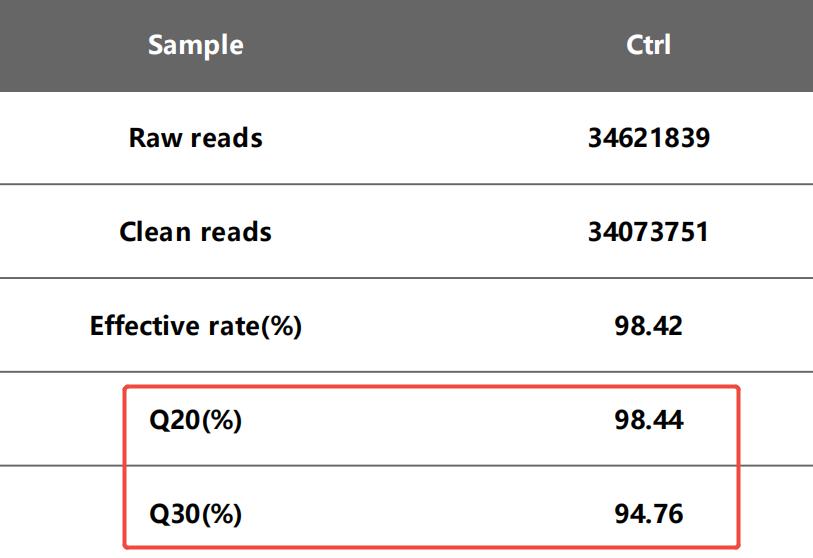
图1 测序数据质量评估
2.数据比对至对应的sgRNA文库
由于sgRNA文库质量、NGS建库以及测序过程中引入突变等因素的影响,所获得的clean reads 中有部分序列无法匹配到对应的sgRNA文库中。为了确保分析的有效性,需要先将clean reads中能匹配到sgRNA文库的reads进行数据比对,从而获取本次CRISPR文库筛选结果中的有效数据(mapped reads)。为了保证测序结果中数据的准确性和可信度,需要分析mapped reads 中的测序深度(mean depth),一般推荐测序深度在300x以上(测序深度 = mapped reads/sgRNA数)。
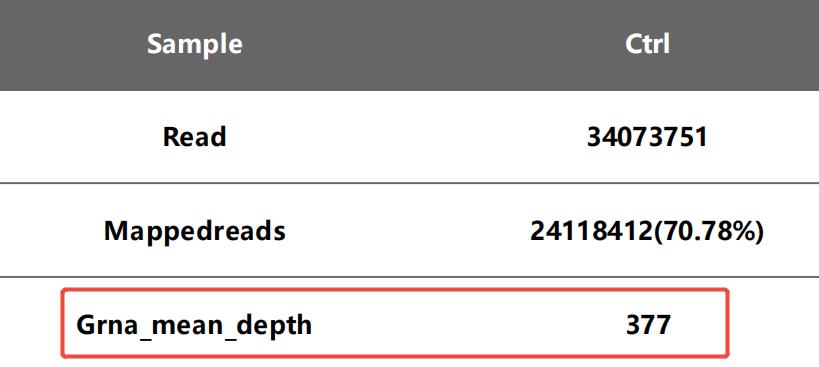
图2 sgRNA测序深度分析
3.差异基因分析
对于CRISPR文库筛选结果,通常使用MAGeCK软件中的RRA(Robust Rank Aggregation)算法[1,2]对实验组和对照组中的sgRNA进行分析,从而找出差异基因。作为一种综合排名算法,RRA算法会对每个基因进行评分和排名,RRA得分越小,排名越靠前,表示该基因是靶基因的可能性越高。此外,在生信分析结果中,会同时分析正向筛选结果和负向筛选结果,正向筛选结果代表该基因在实验组中被显著富集,而负向筛选结果则说明该基因在实验组中显著丢失。
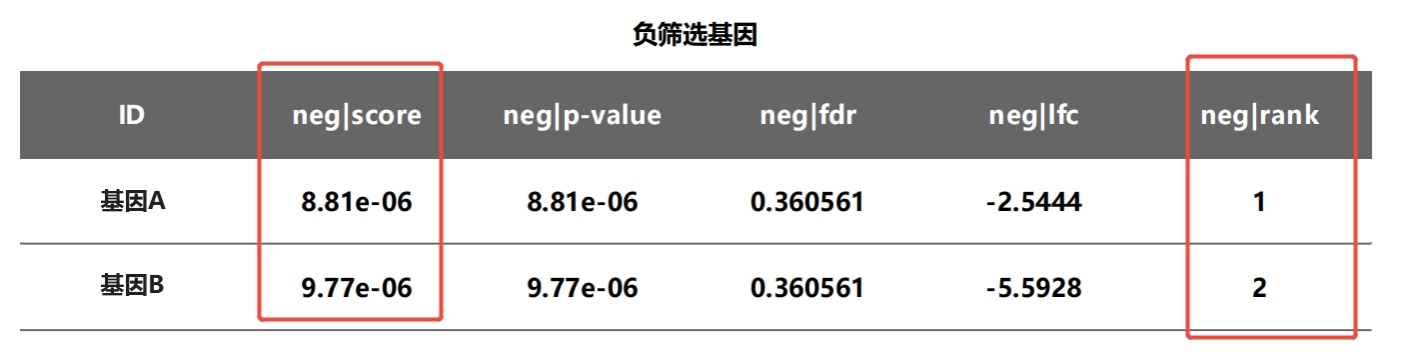
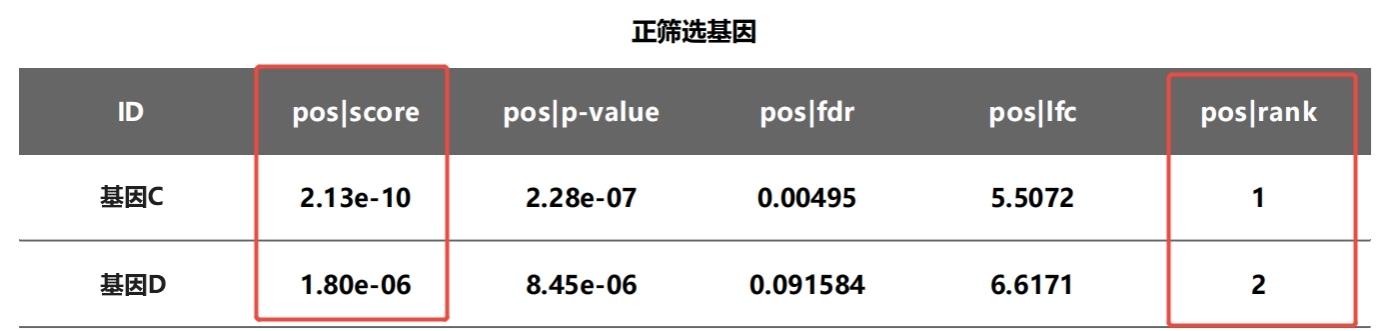
图3 RRA算法分析结果
4.富集分析
筛选出的靶基因会进一步进行GSEA富集分析(图4)以及GO富集分析(图5),揭示差异基因所靶向的信号通路。
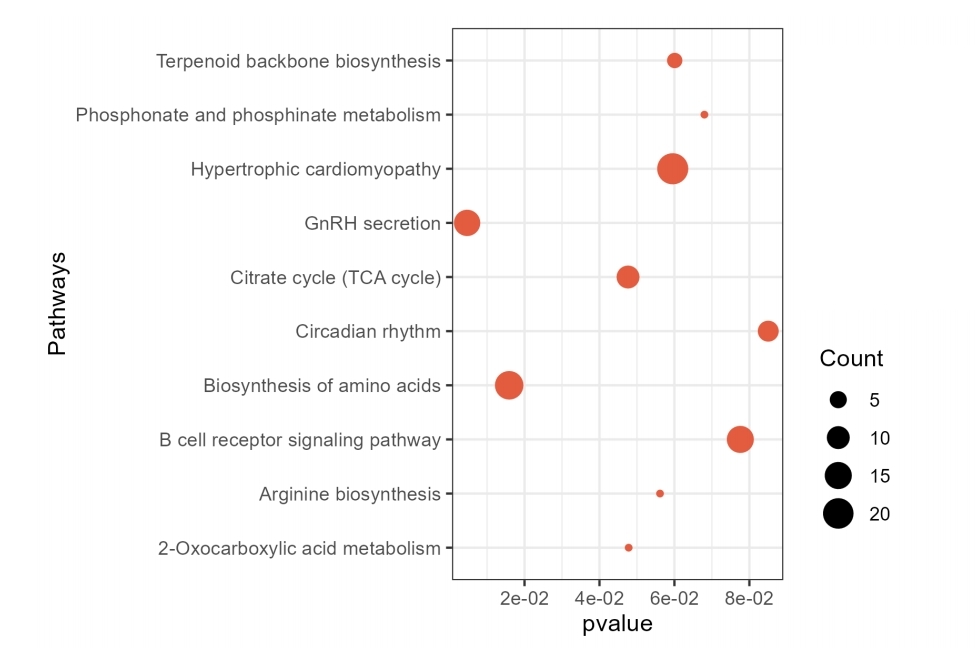
图4 GSEA富集分析
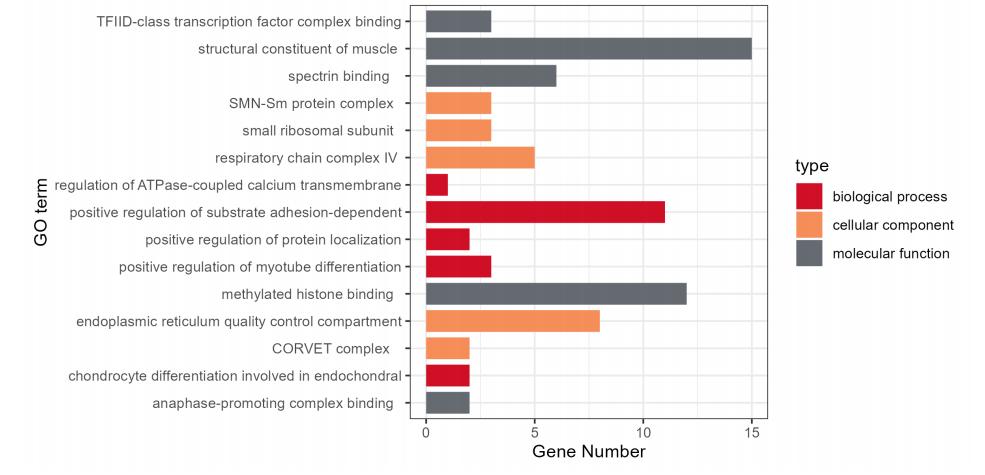
图5 GO富集分析
CRISPR文库潜在靶点分析方法
CRISPR文库作为一种大规模基因筛选的方法,无可避免地会产生一些假阳性的结果,因此在筛选靶基因的过程中,建议多挑选几个基因作为候选基因,并结合下游实验对候选基因进行验证。
1.通过RRA算法rank排名寻找靶点
如前所述,CRISPR文库筛选结果通常使用RRA算法进行分析,排名越靠前的基因,表示该基因是靶基因的可能性越大。在筛选靶基因的过程中,如无法对目的基因进行有效辨别,可以通过筛选排名前20或前30的基因作为候选基因,结合下游基因敲除或过表达实验进行验证。如Liu等人通过RRA算法rank排名找到靶基因Cop1[3]。
![RRA算法排名筛选靶基因Cop1<sup>[3]</sup>](/uploads/allimg/250124/36-250124160I1U4.jpg)
图6 RRA算法排名筛选靶基因Cop1[3]
2.通过p-value、FDR以及LFC这些数值进行筛选
首先,相信大家已经清楚FDR = Q value = adjusted p-value。P-value所代表的是发现某个基因在实验组和对照组中有显著差异的概率,而FDR代表的是错误发现率,也即所有发现中发生了错误所占的比例。简单来说,当p-value < 0.05时,说明该基因在实验组和对照组间存在显著差异的可能性大于95%,当FDR < 0.05时,说明前述判断为真的可能性大于95%。
通常,使用FDR < 0.05作为筛选条件所筛选出的基因是靶基因的可能性更大。然而,由于文库筛选基因数量庞大,往往需要单个基因的p-value < 1*10-7 才可使得FDR < 0.05,单纯使用FDR进行筛选,往往容易遗漏大量真阳性基因。因此,在绝大多数文库筛选案例中,并不会通过FDR,而是p-value来筛选目的基因。
LFC则代表实验组和对照组之间sgRNA差异倍数,当LFC > 1则代表针对某一特定基因实验组中sgRNA数量为对照组的2倍,当LFC > 2则代表该基因实验组中sgRNA数量为对照组的4倍,以此类推。
除了上述提及的通过排名的方式筛选目的基因外,研究者们也可通过p-value和LFC相结合的方法进行筛选潜在靶基因。如Guo等人通过p < 0.01 、LFC ≤ -2的条件筛选到靶基因CDC7[4]。
![结合p-value和LFC筛选出靶基因CDC7<sup>[4]</sup>](/uploads/allimg/250124/36-250124160IJF.jpg)
图7 结合p-value和LFC筛选出靶基因CDC7[4]
希望通过今天小源对 CRISPR 文库分析流程的系统性讲解,能帮助大家消除一些在筛选结果分析及靶点寻找方面的疑惑。后续若大家在实际操作中还有任何问题,欢迎随时与小源交流 。
源井CRISPR文库筛选服务,快至8周筛到新靶点!
还有400+CRISPR文库现货产品(质粒、病毒、Cell Pool),
我们还可提供资质证明,助力拿下国自然,欢迎咨询~
参考文献
[1] Kolde R, Laur S, Adler P, Vilo J. Robust rank aggregation for gene list integration and meta-analysis. Bioinformatics. 2012 Feb 15;28(4):573-80.
[2] Li W, Xu H, Xiao T, Cong L, Love MI, Zhang F, Irizarry RA, Liu JS, Brown M, Liu XS. MAGeCK enables robust identification of essential genes from genome-scale CRISPR/Cas9 knockout screens. Genome Biol. 2014;15(12):554.
[3 ]Wang X, Tokheim C, Gu SS, Wang B, Tang Q, Li Y, Traugh N, Zeng Z, Zhang Y, Li Z, Zhang B, Fu J, Xiao T, Li W, Meyer CA, Chu J, Jiang P, Cejas P, Lim K, Long H, Brown M, Liu XS. In vivo CRISPR screens identify the E3 ligase Cop1 as a modulator of macrophage infiltration and cancer immunotherapy target. Cell. 2021 Oct 14;184(21):5357-5374.e22.
[4] Deng L, Yang L, Zhu S, Li M, Wang Y, Cao X, Wang Q, Guo L. Identifying CDC7 as a synergistic target of chemotherapy in resistant small-cell lung cancer via CRISPR/Cas9 screening. Cell Death Discov. 2023 Feb 2;9(1):40.
 CRISPR基因编辑技术服务
CRISPR基因编辑技术服务
 红棉 · 基因敲除计划
红棉 · 基因敲除计划












![RRA算法排名筛选靶基因Cop1<sup>[3]</sup>](/uploads/allimg/250124/36-250124160I1U4.jpg)
![结合p-value和LFC筛选出靶基因CDC7<sup>[4]</sup>](/uploads/allimg/250124/36-250124160IJF.jpg)











