单细胞CRISPR文库的应用,你知道几种?

CRISPR文库筛选技术能够通过特定的细胞表型(如细胞生长、迁移、耐药性以及抗原抗体的表达等)筛选富集靶细胞,并结合NGS测序找出与该表型相关的潜在靶点。然而,对于无明显表型变化或难以通过表型进行细胞筛选的研究,仅通过CRISPR文库筛选难以找出潜在靶点。单细胞测序技术能够实现单个细胞的转录组测序,从分子层面判别细胞与细胞之间的差异;结合CRISPR文库,单细胞CRISPR文库技术能同时获取多个不同突变类型细胞的转录组数据,在分子水平检测细胞之间的差异。
单细胞CRISPR文库技术简介
单细胞CRISPR文库技术最初由Broad研究所Dixit团队开发(命名为Perturb-Seq),通过将CRISPR文库与单细胞测序技术结合,可实现同时检测多个不同细胞基因敲除/激活造成的基因转录本变化情况。
与传统的单细胞测序技术相比,单细胞CRISPR文库技术的难点在于需要将细胞中的sgRNA信息与单细胞测序数据一一对应;然而由于sgRNA骨架上不含有polyA序列,常规的单细胞建库方法无法直接获取单个细胞中的sgRNA信息。为了解决这一问题,Dixit等人在慢病毒载体中添加一个barcode序列,并与sgRNA序列一一对应,通过单细胞测序识别barcode序列后,即可将sgRNA信息与细胞测序结果相匹配[1](图1)。
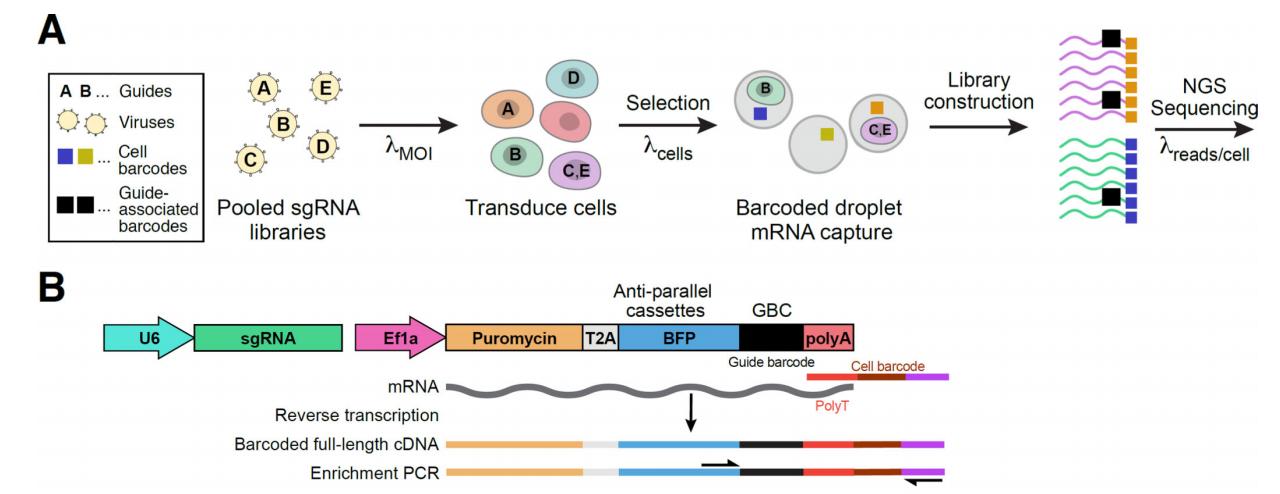
图1 Perturb-Seq 测序原理[1]
各研究团队又在此基础上对此方法进行了优化,开发出了CRISP-Seq[2]、CROP-Seq[3]以及目前最常用的Direct capture perturb-seq[4]等方法;并且,Direct capture perturb-seq所提供的5端测序方案(图2 A)中,无需对CRISPR文库骨架进行改造即可实现单细胞CRISPR文库测序,对相关研究提供了极大便利。
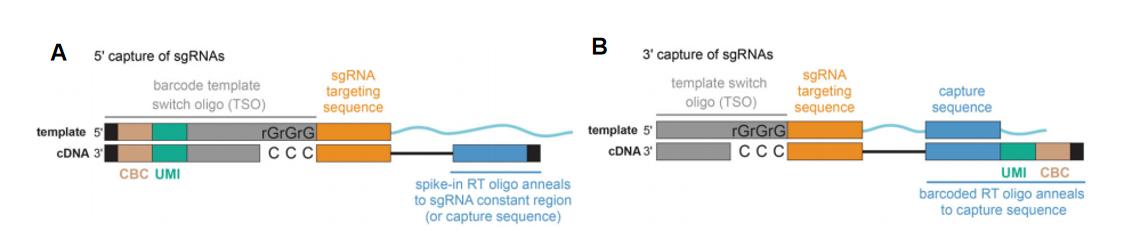
图2 Direct capture perturb-seq测序原理[4]
接下来,让小源为大家介绍一下单细胞CRISPR文库技术适用于哪些研究方向吧!
单细胞CRISPR文库技术应用
1. 通过转录本变化情况筛选靶点
如前所述,单细胞CRISPR文库技术能够克服CRISPR文库筛选技术对于表型的依赖,即使没有可用于筛选的表型特征(如精神类疾病),单细胞CRISPR文库技术也能用于寻找潜在靶点。
22q11.2染色体缺失综合征是一种由于染色体部分缺失所导致的遗传性疾病,在该段染色体中共含有37个基因,其中29个在大脑前额叶皮质中表达,临床上患者通常表现为精神分裂症或自闭症。为了深入研究其中哪些基因是致病的关键基因,研究者构建了靶向这29个基因的CRISPR文库,通过流式分选神经细胞以及单细胞测序分析不同细胞间的转录组变化(图3),他们发现Dgcr8、Dgcr14、Gnb1l和Ufd1l 基因敲除的各类型神经细胞中,其转录本均发生明显变化(图4 C)。通过比对这些基因敲除后的神经细胞与22q11.2染色体缺失小鼠模型中神经细胞的转录本差异,研究者们也进一步确认Dgcr8和Gnb1l基因缺失与该疾病的发展具有明显相关性[5]。
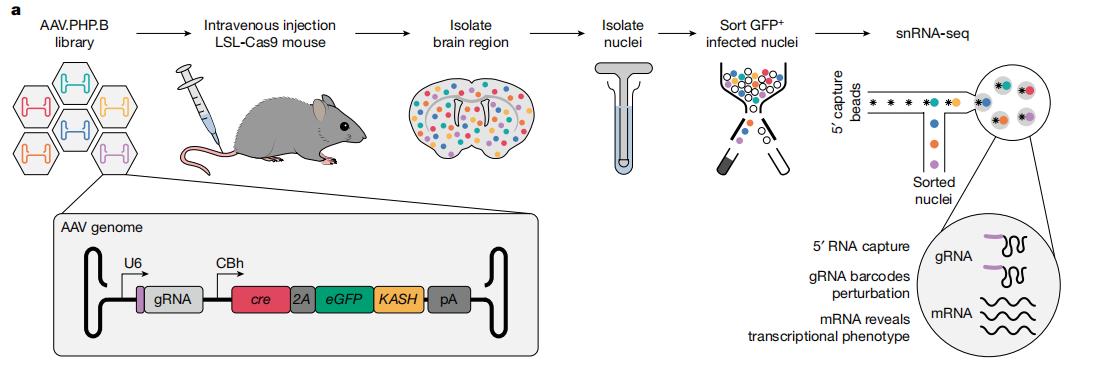
图3 22q11.2染色体缺失综合征相关基因单细胞CRISPR文库技术路线[5]
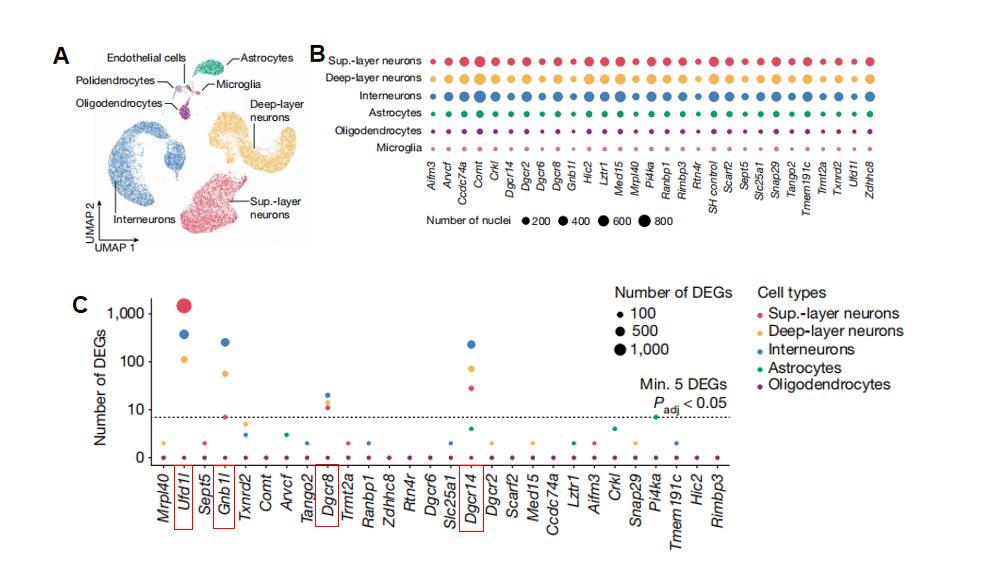
图4 不同基因敲除细胞表达谱变化[5]
2. 分析复合物中不同基因/亚基的功能
哺乳动物SWI/SNF复合物(BAF)是由29个基因调控表达的10—15个亚基组合而成,该复合物对DNA的可及性以及基因的表达具有重要作用,然而复合物中不同亚基对调控基因表达的贡献仍不明晰。Cigall 等人针对该复合物中的28个基因设计了CRISPR敲除文库,通过慢病毒转染MOLM-13细胞,并结合流式分选以及单细胞测序技术进行分析后,揭示了该复合物中不同亚基对DNA可及性以及基因表达的作用。由于BAF复合物存在3种不同的形式(cBAF、PBAF和ncBAF),作者对这三种不同形式的复合物对应的基因表达谱分别进行分析,发现PBAF对基因表达的影响最小,而cBAF对发育和分化相关基因的表达调控影响最大[6]。
![5 SWI/SNF复合物单细胞CRISPR文库技术路线<sup>[6]</sup>](/uploads/allimg/250312/36-25031215261MQ.jpg)
图5 SWI/SNF复合物单细胞CRISPR文库技术路线[6]
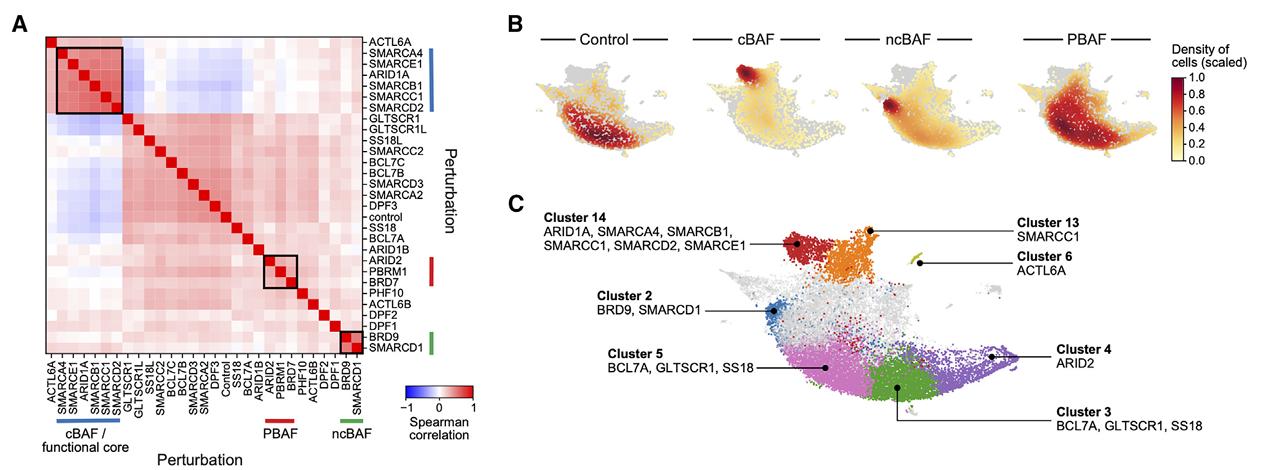
图6 不同基因、复合物亚型表达谱变化[6]
3. 比较不同细胞类群sgRNA差异寻找细胞分化相关基因
CD8+毒性T细胞(CTL)是抗肿瘤反应中的主要免疫细胞,其中,Tex细胞是CTL的主要群体,能直接杀伤肿瘤细胞,但是Tex增殖能力有限;相比而言,Tex细胞的前体细胞Tpex能分化产生大量的Tex细胞。然而,Tpex到Tex细胞分化的基因调控网络尚未被完全理解。为探究上述问题,迟洪波团队构建了靶向180个细胞命运决定相关转录因子的CRISPR敲除文库CTL细胞, 在将文库细胞转入黑色素瘤小鼠一周后,通过流式分选出剩余细胞,并进行单细胞测序。通过细胞分化相关基因对细胞进行分群以及pesudotime分析后,研究团队将细胞分为逐级分化的4个类群,即Tpex1、Tpex2、Tex1和Tex2。比较分析不同细胞类群中sgRNA富集信息,团队发现Ikzf1基因能够促使Tpex1西部那退出静息状态,促进Tpex1细胞分化为Tpex2细胞;同时,Ets1基因则负向调控Tpex2分化为Tex1细胞,抑制Tpex细胞的分化;而Tex1细胞分化为Tex2细胞则依赖于Rbpj基因的激活[7]。总的来说,该团队通过单细胞CRISPR文库技术发现并建立了Tpex细胞分化为Tex细胞过程中的关键调控通路。
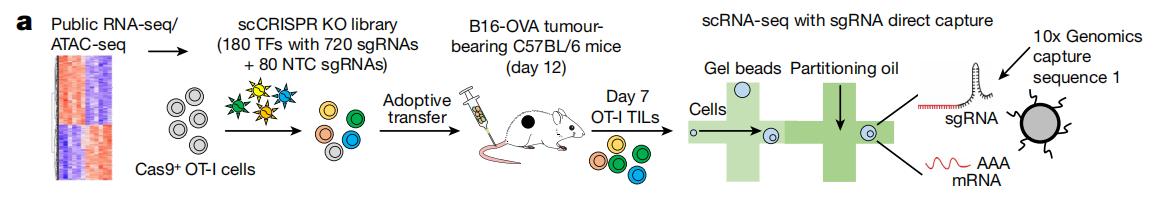
图7 单细胞CRISPR文库筛选细胞分化相关转录因子[7]
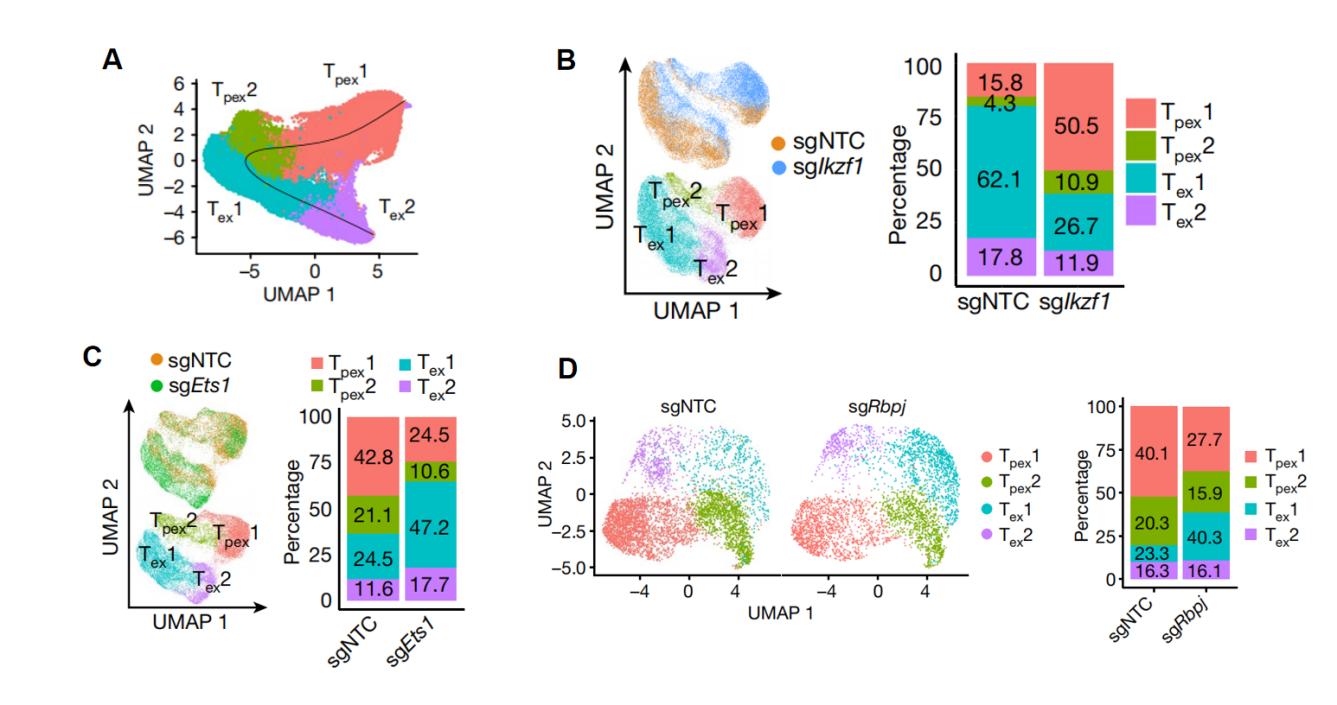
图8 Pesudotime分析以及各细胞群中sgRNA富集信息[7]
总结
单细胞CRISPR文库技术是利用CRISPR文库同时向多个细胞引入不同突变,再结合单细胞测序获取这些具有不同突变细胞转录本数据进行分析的方法。与传统的单细胞测序技术相比,单细胞CRISPR文库技术能同时获取多个不同基因敲除细胞的转录本信息[5];结合下游单细胞数据分析方法,不仅能实现细胞分群、Pesudotime分析,还能结合sgRNA信息对细胞进行分类或比较不同细胞群体之间的sgRNA差异,以此寻找靶点[6-7]。
源井生物可提供400种文库现货,现在CRISPR文库质粒低至199元,400种Cell Pool低至8888元,想筛就筛!我们还可提供单细胞CRISPR文库的一站式服务,多种交付方式满足不同科研需求。
参考文献
[1]Dixit A, Parnas O, Li B, Chen J, Fulco CP, Jerby-Arnon L, Marjanovic ND, Dionne D, Burks T, Raychowdhury R, Adamson B, Norman TM, Lander ES, Weissman JS, Friedman N, Regev A. Perturb-Seq: Dissecting Molecular Circuits with Scalable Single-Cell RNA Profiling of Pooled Genetic Screens. Cell. 2016 Dec 15;167(7):1853-1866.e17.
[2]Jaitin DA, Weiner A, Yofe I, Lara-Astiaso D, Keren-Shaul H, David E, Salame TM, Tanay A, van Oudenaarden A, Amit I. Dissecting Immune Circuits by Linking CRISPR-Pooled Screens with Single-Cell RNA-Seq. Cell. 2016 Dec 15;167(7):1883-1896.e15.
[3]Datlinger P, Rendeiro AF, Schmidl C, Krausgruber T, Traxler P, Klughammer J, Schuster LC, Kuchler A, Alpar D, Bock C. Pooled CRISPR screening with single-cell transcriptome readout. Nat Methods. 2017 Mar;14(3):297-301.
[4]Replogle JM, Norman TM, Xu A, Hussmann JA, Chen J, Cogan JZ, Meer EJ, Terry JM, Riordan DP, Srinivas N, Fiddes IT, Arthur JG, Alvarado LJ, Pfeiffer KA, Mikkelsen TS, Weissman JS, Adamson B. Combinatorial single-cell CRISPR screens by direct guide RNA capture and targeted sequencing. Nat Biotechnol. 2020 Aug;38(8):954-961.
[5]Santinha AJ, Klingler E, Kuhn M, Farouni R, Lagler S, Kalamakis G, Lischetti U, Jabaudon D, Platt RJ. Transcriptional linkage analysis with in vivo AAV-Perturb-seq. Nature. 2023 Oct;622(7982):367-375.
[6]Otto JE, Ursu O, Wu AP, Winter EB, Cuoco MS, Ma S, Qian K, Michel BC, Buenrostro JD, Berger B, Regev A, Kadoch C. Structural and functional properties of mSWI/SNF chromatin remodeling complexes revealed through single-cell perturbation screens. Mol Cell. 2023 Apr 20;83(8):1350-1367.e7.
[7]Zhou P, Shi H, Huang H, Sun X, Yuan S, Chapman NM, Connelly JP, Lim SA, Saravia J, Kc A, Pruett-Miller SM, Chi H. Single-cell CRISPR screens in vivo map T cell fate regulomes in cancer. Nature. 2023 Dec;624(7990):154-163.
 CRISPR基因编辑技术服务
CRISPR基因编辑技术服务
 红棉 · 基因敲除计划
红棉 · 基因敲除计划










![5 SWI/SNF复合物单细胞CRISPR文库技术路线<sup>[6]</sup>](/uploads/allimg/250312/36-25031215261MQ.jpg)















